Nature子刊:山东大学张磊/赵国平团队开发AI大模型,用于发现抗菌肽,对抗多重耐药菌
来源:网络 2025-10-11 10:34
该研究开发了一个预训练的蛋白质大语言模型(pre-trained protein LLM)——ProteoGPT,用于挖掘和生成针对多重耐药菌的新型抗菌肽。
世界卫生组织(WHO)曾发一份多重耐药菌名单,统称为 ESKAPE,代表了六种最棘手、最常见的多重耐药细菌,名单之首是耐碳青霉烯类鲍曼不动杆菌(CRAB)。碳青霉烯类抗生素是所有其他治疗手段都失败时的“最后一道防线”,但其极易受到抗生素耐药性的出现和传播的影响。鉴于这一紧迫问题,人们越来越关注抗菌肽(AMP)作为传统抗生素的有前景替代品。
与传统抗生素相比,抗菌肽(AMP)因其广谱活性、快速杀菌机制以及诱导耐药性的可能性较小,成为很有前景的抗生素替代品。发现针对临床多重耐药菌的新型抗菌肽,对于应对持续的抗生素耐药危机至关重要。
2025 年 10 月 3 日,山东大学齐鲁医学院张磊教授、赵国平教授团队在 Nature 子刊 Nature Microbiology 上发表了题为:A generative artificial intelligence approach for the discovery of antimicrobial peptides against multidrug-resistant bacteria 的研究论文。
该研究开发了一个预训练的蛋白质大语言模型(pre-trained protein LLM)——ProteoGPT,用于挖掘和生成针对多重耐药菌的新型抗菌肽,从而能够高效且广泛地探索抗菌肽空间,以应对临床超级细菌。

大语言模型(LLM)已成为提升自然语言理解能力的变革力量,是迈向通用人工智能(Artificial General Intelligence,AGI)的重要一步。通常来说,大语言模型指的是基于 Transformer 架构构建的人工智能模型,拥有数亿(甚至数十亿)个可训练参数,并在海量文本语料库上进行训练。然而,现有的通用大语言模型在处理科学数据(例如分子、蛋白质和基因)时往往力不从心。为了促进对科学语言的理解,专门针对不同科学领域和学科定制的科学大语言模型应运而生。
在这项最新研究中,研究团队建立了一个预训练的蛋白质大语言模型(pre-trained protein LLM)——ProteoGPT,并将其进一步开发成多个专业化的子 LLM,以构建一个顺序流程。该流程能够对数亿种多肽序列进行快速筛选,确保其具有强大的抗菌活性,并将细胞毒性风险降至最低。通过迁移学习,研究团队赋予了 ProteoGPT 不同的特定领域知识,从而在统一的方法框架内实现了抗菌肽的高通量挖掘和生成。
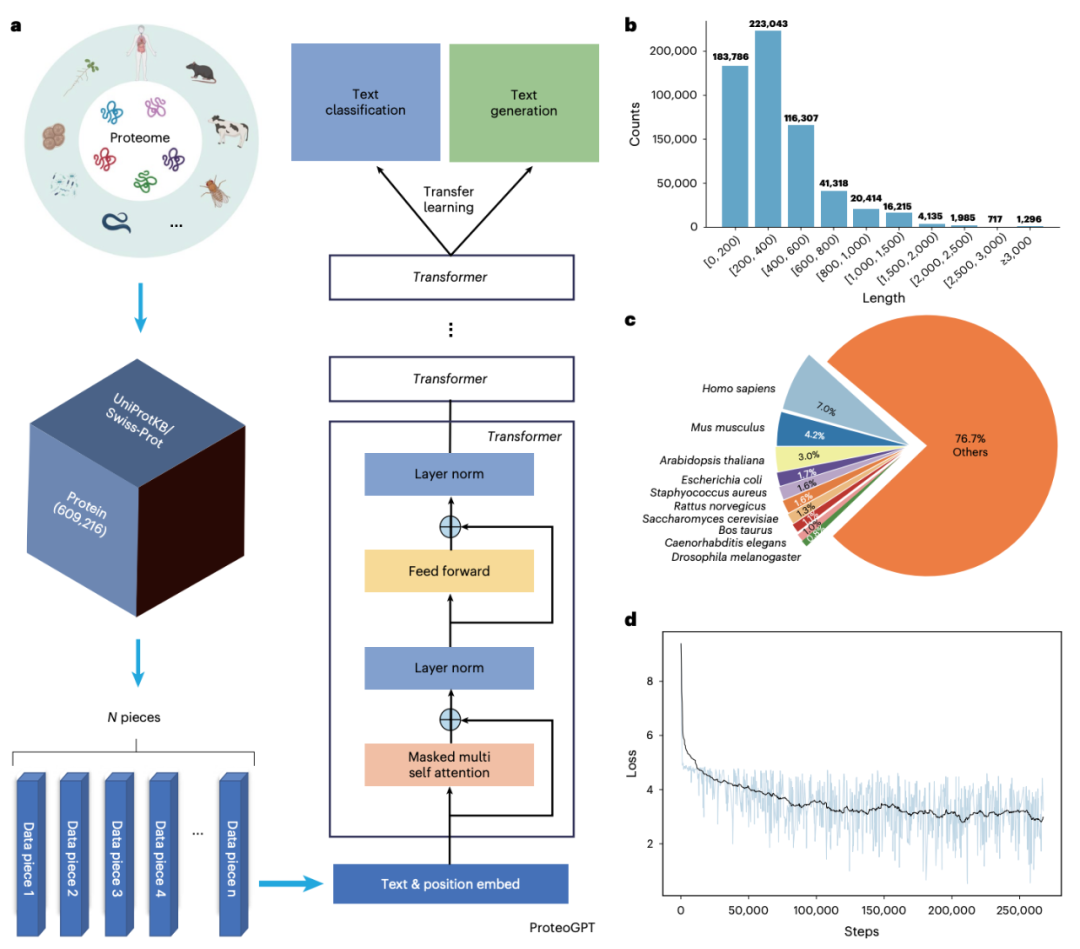
值得注意的是,在体外实验中,无论是挖掘筛选到的还是生成的抗菌肽,对从重症监护病房分离出的耐碳青霉烯类鲍曼不动杆菌(CRAB)和耐甲氧西林金黄色葡萄球菌(MRSA)的耐药性发展都表现出较低的易感性。在小鼠大腿感染的体内动物模型中,这些抗菌肽也显示出与临床使用的抗生素相当甚至更优的治疗效果,且不会造成器官损伤和破坏肠道微生物群。这些抗菌肽的作用机制包括破坏细胞质膜和膜去极化。
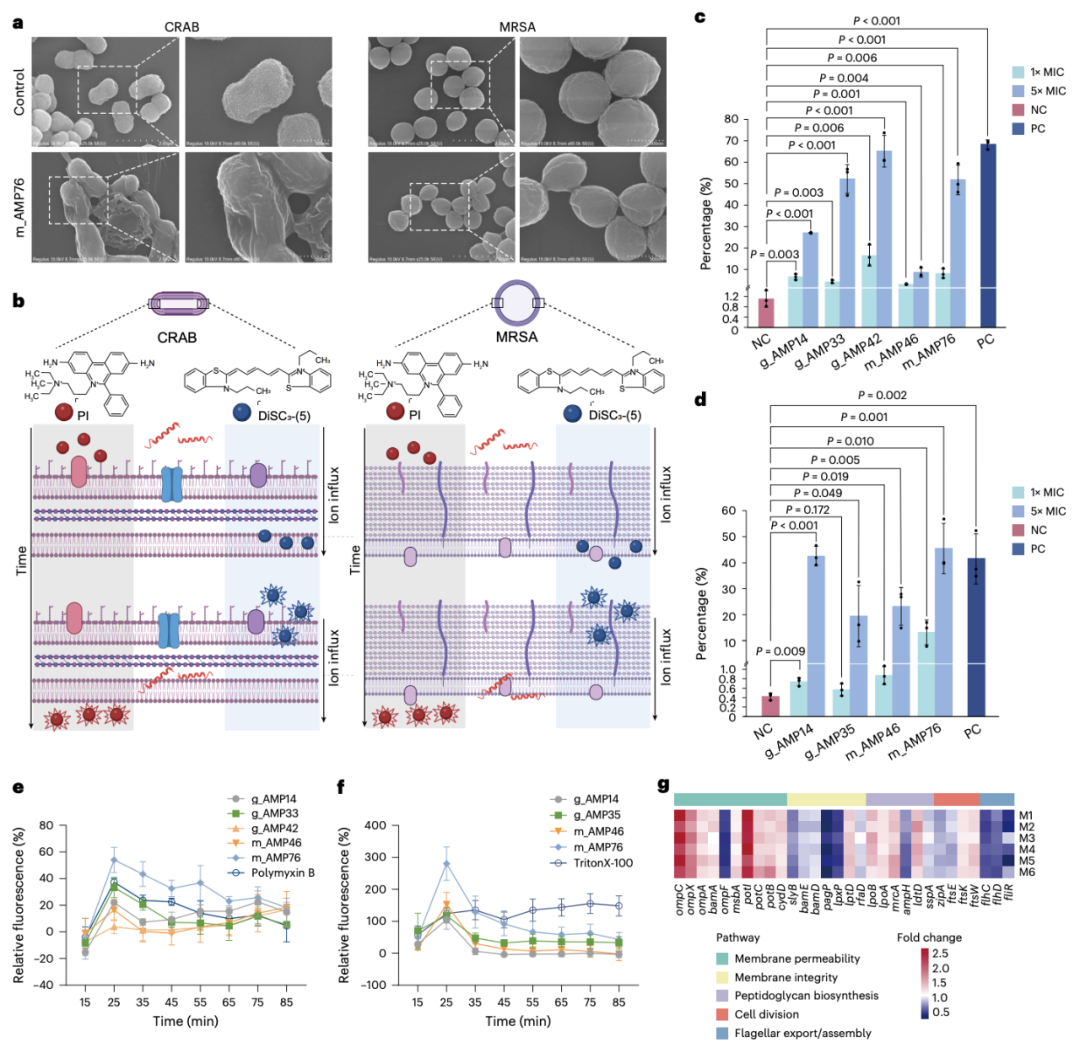
总的来说,该研究提出了一种基于大语言模型(LLM)的生成式人工智能方法,能够利用数据挖掘和文本生成策略,实现高效且安全的抗菌肽的高通量发现,从而能够高效且广泛地探索抗菌肽空间,以应对临床超级细菌。
版权声明 本网站所有注明“来源:生物谷”或“来源:bioon”的文字、图片和音视频资料,版权均属于生物谷网站所有。非经授权,任何媒体、网站或个人不得转载,否则将追究法律责任。取得书面授权转载时,须注明“来源:生物谷”。其它来源的文章系转载文章,本网所有转载文章系出于传递更多信息之目的,转载内容不代表本站立场。不希望被转载的媒体或个人可与我们联系,我们将立即进行删除处理。


